fNIRS
Espectroscopia do infravermelho próximo.
A espectroscopia do infravermelho próximo em funcionamento - fNIRs é uma tecnologia de neuroimagem para mapear o córtex humano, que desde 1977, é empregada para o mapeamento do cérebro de humanos e em animais através de sua oxigenação (Jobsis, 1977). Os tecidos humanos são relativamente transparentes à luz na janela espectral NIR, que é absorvida por compostos pigmentados (cromóforos) ou dispersa em tecidos (Ferrari, 2012). A luz NIR é capaz de penetrar nos tecidos humanos, uma vez que o fator dominante no seu transporte de tecido é a dispersão. A imagem em infravermelho próximo ou a imagem óptica difusa são utilizadas para detectar mudanças simultâneas nas propriedades ópticas do córtex humano a partir de múltiplos locais de medição e exibe os resultados na forma de um mapa ou imagem em uma área específica.
Atividade cerebral está associada a um número de eventos fisiológicos, alguns associados a mudanças nas propriedades ópticas do tecido cerebral e podem ser avaliadas por técnicas ópticas. As principais vantagens dos métodos ópticos incluem custo relativamente baixo, a especificidade bioquímica, a resolução temporal (na faixa de milissegundos), o potencial para medir simultaneamente os eventos intracelulares e intravasculares e a facilidade com que os dispositivos podem ser transportados (Ferrari, 2012).
Segundo Delpy (10997), utiliza-se níveis seguros de luz laser (com comprimentos de onda entre 650 e 1000 nm) para inferir a variação do nível de oxigenação do tecido cerebral de forma não invasiva, que penetram no tecido biológico e atingem o córtex, permitindo analisar a oxi-hemoglobina (O2Hb), deoxi-hemoglobina (HHb) e hemoglobina total (tHb; tHb = O2Hb + HHb) do sangue cerebral. Muitos outros cromóforos ainda absorvem a luz mas apenas essas são de importância clínica e exibem absorção dependente da oxigenação. Através delas pode-se buscar um melhor entendimento do acoplamento neuro-vascular, das flutuações neurofisiológicas e da hemodinâmica cerebral.
A técnica NIRs utiliza:
- Diodo laser e/ou fontes de luz de diodos emissores de luz que abrangem a janela óptica entre 650 e 1000 nm
- Fibra óptica flexível para transportar a luz NIR da fonte e detector tecidos
Três tipos de NIRS diferentes são usadas, cada uma baseada em um tipo específico de iluminação:
A modalidade CW ( ou ondas contínuas) que, com base na constante iluminação do tecido, mede a atenuação da luz através da cabeça. Essa modalidade mede apenas as alterações de oxigenação de O2Hb e HHb mas em relação às outras técnicas, oferece as vantagens de baixo custo e facilidade de transporte.
O método de freqüência-domínio (FD) que, iluminando a cabeça com luz modulada por intensidade, mede a atenuação e o atraso de fase da luz emergente.
A técnica do domínio do tempo (TD) que, iluminando a cabeça com pulsos de luz curtos, detecta a forma do pulso após a propagação através dos tecidos.
As modalidades FD e TD oferecem a possibilidade de caracterizar as propriedades ópticas dos tecidos (absorção e coeficientes de espalhamento reduzidos), dos quais é possível recuperar as concentrações absolutas de O2Hb e HHb.
As fibras ópticas são muito adequadas para qualquer posição e postura da cabeça. As medições de NIRS podem ser realizadas em ambientes naturais sem necessidade de restrição ou sedação e a profundidade adequada da penetração da luz NIR (quase a metade da distância do detector fonte) pode ser alcançada usando uma distância do detector em torno de 3cm. A seleção da distância ideal do detector de fonte depende da intensidade da luz NIR e do comprimento de onda, da idade do sujeito e a região da cabeça medida (Ferrari, 2012).
À medida que a luz injetada se difunde em todas as direções dentro dos tecidos da cabeça (couro cabeludo, crânio e espaço subaracnóideo preenchido com líquido cefalorraquidiano) tanto antes como depois de passar pelo tecido cerebral, a sensibilidade de cada par de fontes-detectores exibe uma distribuição espacial da luz NIR através das diferentes camadas com formato similar ao de uma banana (duas extremidades estreitas e uma curva para dentro em direção ao centro).
A atenuação relativamente alta da luz NIR no tecido é devida à hemoglobina principal do cromóforo (a proteína dos glóbulos vermelhos que faz o transporte de oxigênio) localizada em pequenos vasos (
Conforme apresentado por (Mesquita, 2008), O tecido biológico é altamente espalhador de luz e possui coeficientes de absorção relativamente baixos na região do infravermelho próximo do espectro eletromagnético. Estas propriedades permitem a penetração da luz no tecido e sua conseqüente propagação por alguns milímetros, através dos coeficientes de absorção
Durante ativação cerebral (Mesquita2008), as variações nas concentrações de O2Hb e HHb resultam em variações na absorção da luz dentro do tecido. Para medidas hemodinâmicas funcionais típicas, assume-se que essas variações são pequenas, insuficientes para perturbar o caminho da luz através do tecido podendo ser generalizada através da Lei de Beer-Lambert:
onde,
No entanto, essa equação só é válida na ausência de espalhamento. Como o caminho médio da luz depende das propriedades de espalhamento do tecido, usa-se a lei de Beer-Lambert modificada, (MBLL, do inglês ) para contabilizar estes efeitos e levar em conta a propagação difusiva da luz, que inclui um fator de caminho diferencial adicional (
onde a variação de concentração do n-ésimo cromóforo,
Os experimentos de NIRS, no entanto, medem apenas as variações relativas de oxigenação do tecido. Os valores absolutos não são disponíveis, principalmente levando-se em consideração as diferentes estruturas cerebrais pelas quais a luz atravessa. Outras limitações também são: (a) a interferência da espessura do crânio (medidas cerebrais) ou da espessura do tecido adiposo (medidas musculares); (b) a contribuição controversa desconhecida da mioglobina no sinal muscular NIRS; (c) o efeito do volume sanguíneo muda no comprimento do tecido e, consequentemente no volume de amostra observado; (d) a dificuldade de prever o quanto de uma mudança de sinal NIRS observada é devido ao fluxo sanguíneo cerebral vs. couro cabeludo, ou (e) mudanças simultâneas no fluxo e no volume. Ainda assim, dado o fato de que a fração do volume de sangue arterial é de aproximadamente 30% no cérebro humano, a técnica NIRS oferece a possibilidade de obter informações sobre alterações de oxigenação que ocorrem dentro do compartimento venoso, sendo uma técnica bastante eficaz para detectar zonas de ativação cerebral.
Ao longo do tempo, o que se altera nos dados de fNIRS é a variação de oxyhemoglobina e deoxyhemoglobina.
No caso da coleta, o canal é o caminho entre o emissor e o detector. A montagem é escolhida de forma a varrer as regiões cerebrais as quais se pretende estudar (aprox 3cm).
http://www.youtube.com/watch?v=CJIZkZ2ol5A
E também:
http://www.youtube.com/watch?v=3LMXiDzcANQ
Realizando a leitura dos dados de fNIRS no R:
# Os dados já foram preprocessados
# - PCA para correcao de movimento
# - retirar outliers
# - filtro temporal: passa-banda (0.001-0.1Hz)
#Leitura de dados
OXY = read.table("./data/Oxy781HZ30sBlock.txt", header=F)
DEOXY = read.table("./data/Deoxy781HZ30sBlock.txt", header=F)Analisando o tamanho do arquivo e a taxa de amostragem:
# Taxa de amostragem do equipamento
HZ = 7.81
dim(OXY)## [1] 2660 20dim(DEOXY)## [1] 2660 20# Logo, temos de tempo de coleta:
tempo = dim(OXY)/HZ
tempo## [1] 340.588988 2.560819ts.plot(OXY[, 1])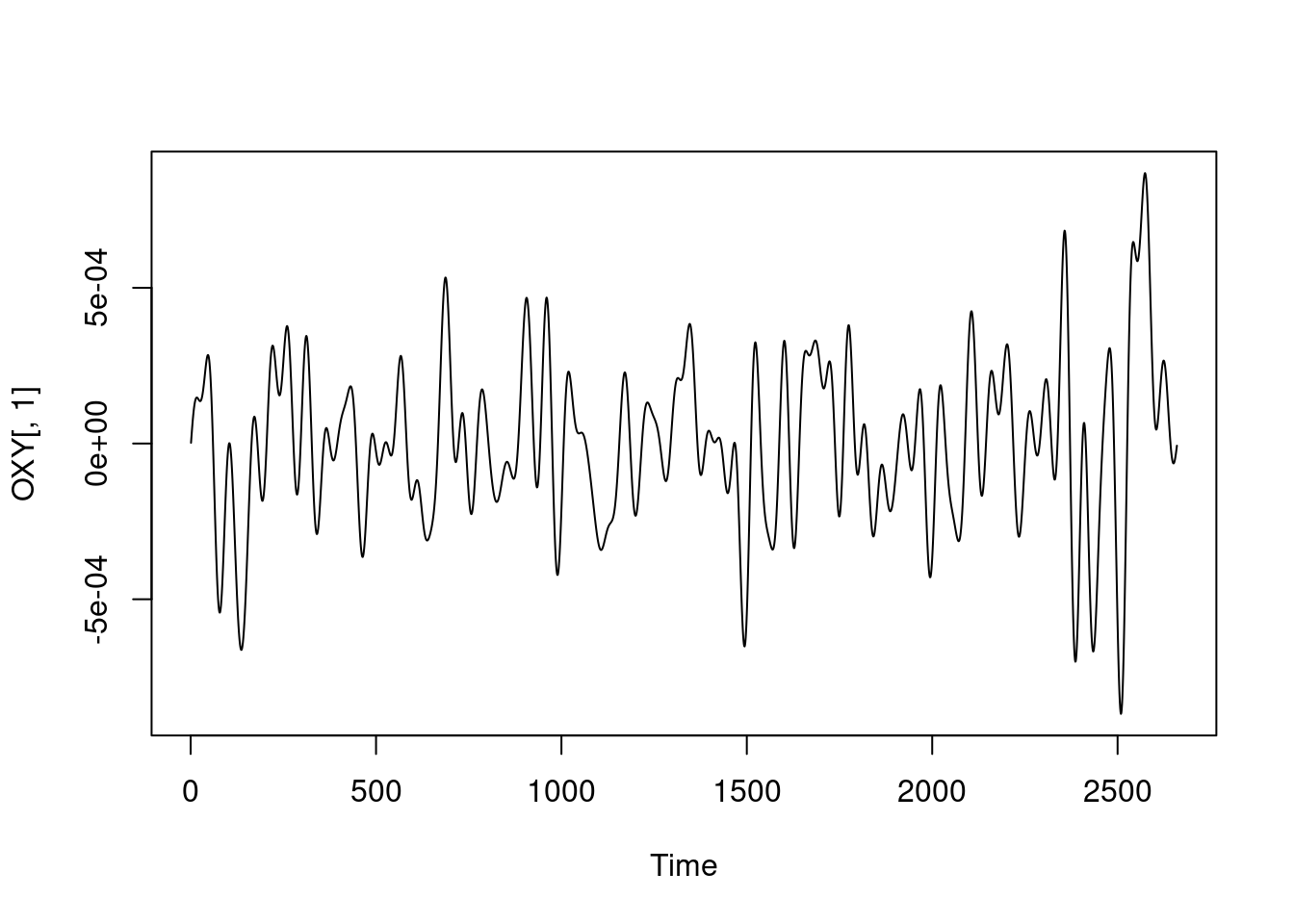
ts.plot(DEOXY[, 1])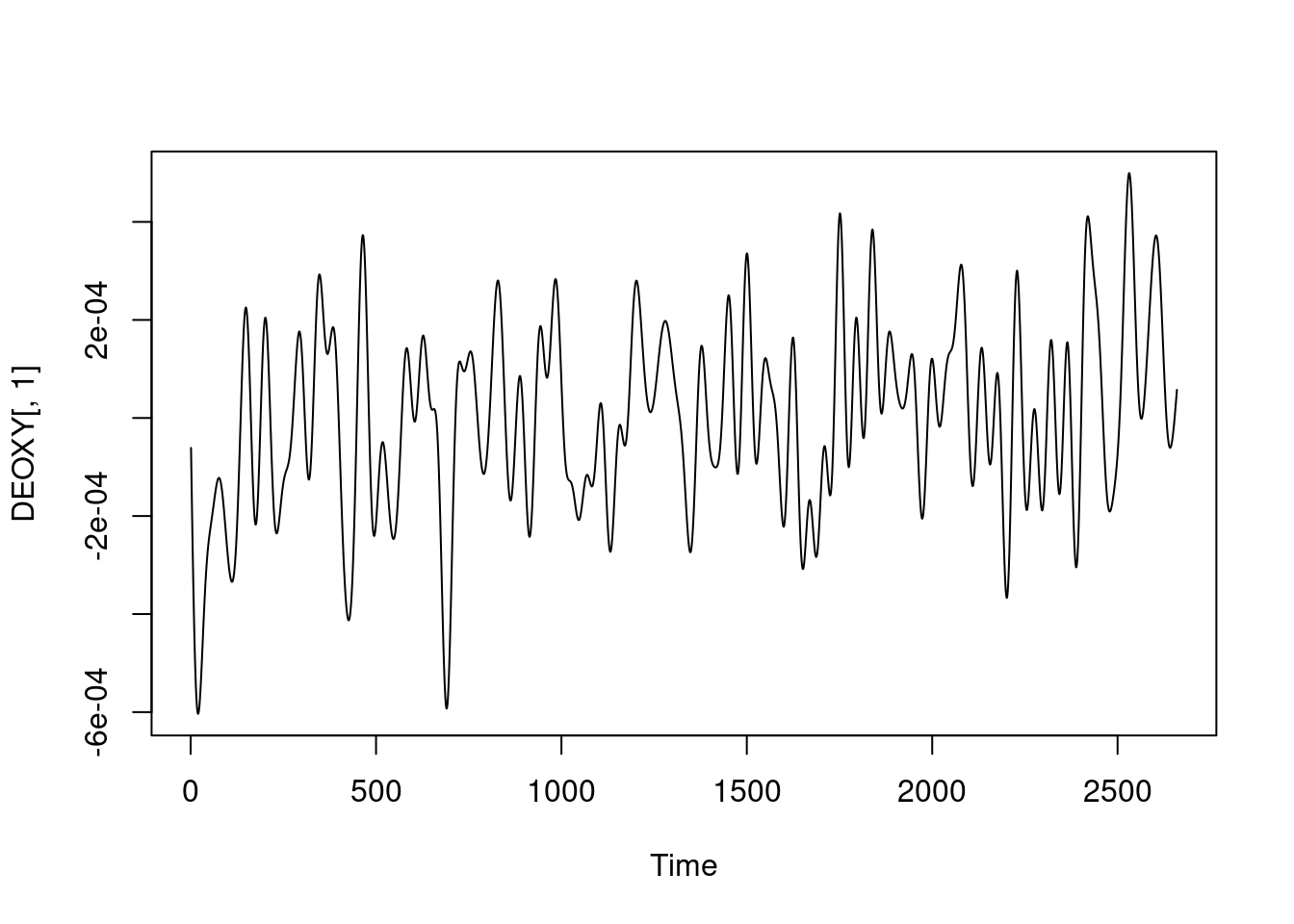
#Tarefa block-design - 30s
COND = 330Considerando uma tarefa que intercala Repouso e tarefa de 30s, ou seja nas seguintes condições (convertendo para frames): R: 1:234 T: 235:469 R: 470:703 T: 704:937 R: 938:1172 T: 1173:1406 R: 1407:1640 T: 1641:1874 R: 1875:2109 T: 2110:2343 R: 2344:2577
#Tarefa block-design - 30s
COND = array(0, 2577) #2577 para os primeiros 330s
COND[235:469] = 1
COND[704:937] = 1
COND[1173:1406] = 1
COND[1641:1874] = 1
COND[2110:2343] = 1
ts.plot(COND)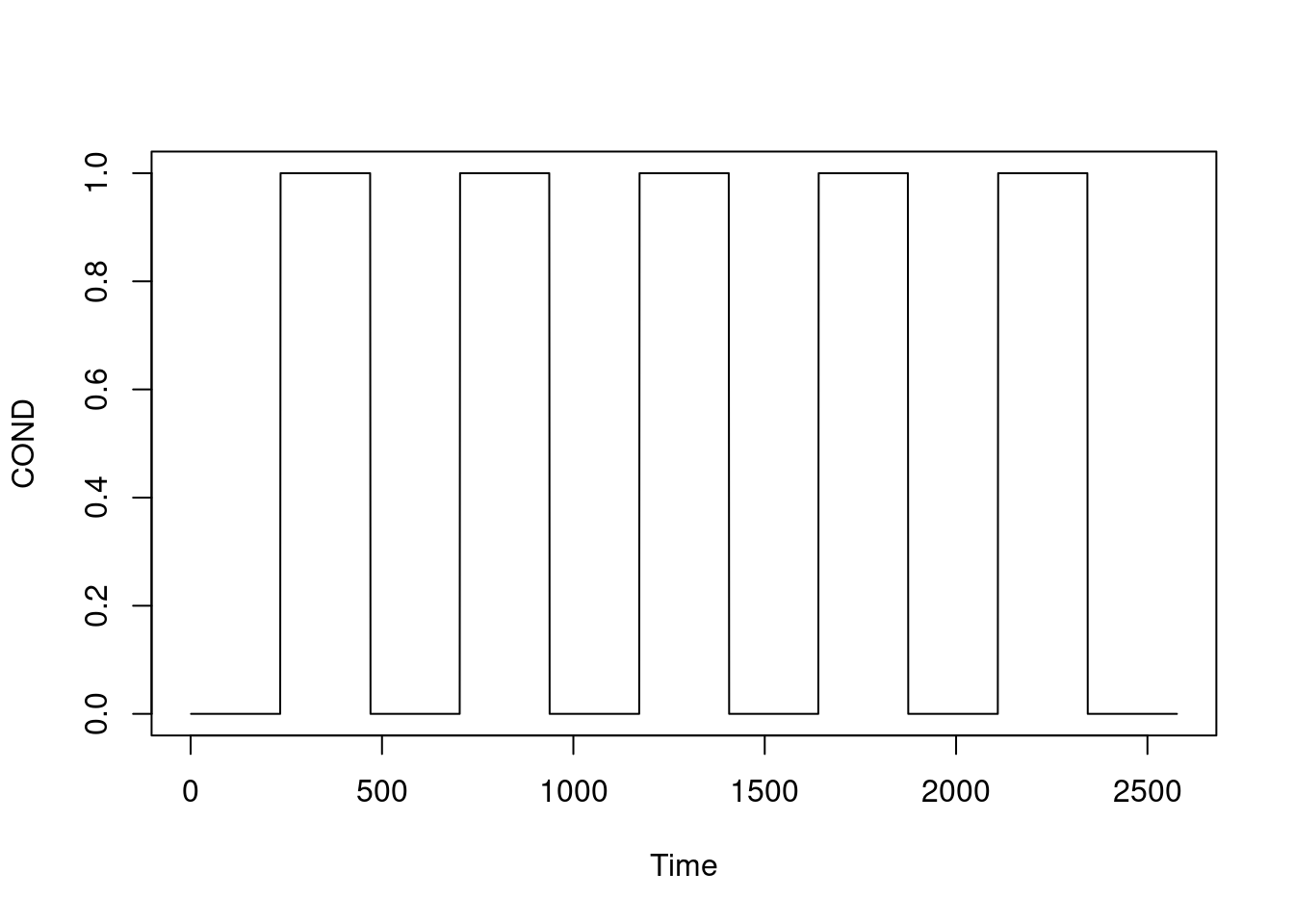
Considerando a função glover para convolução pela HRF canônica, para verificar como a Oxyhemoglobina se comporta.
Para prever o desenho experimental pela HRF, similar às aulas de ressonância:
glover = function(HZ){
a1 = 6
a2 = 12
b1 = 0.9
b2 = 0.9
d1 = 5.4
d2 = 10.8
c = 0.35
x = seq(0, 30, 1/HZ) # HZ is the Sampling Rate (Heartz)
glover1 = ((x/d1)^a1)*exp((-x+d1)/b1)
glover2 = ((x/d2)^a2)*exp((-x+d2)/b2)
G = glover1-c*glover2
return(G)
}
#HRF canonica
HRF = glover(HZ)
#Convolucao da condicao da tarefa pela HRF
X = filter(COND, HRF, method="convolution", side=1)
ts.plot(X)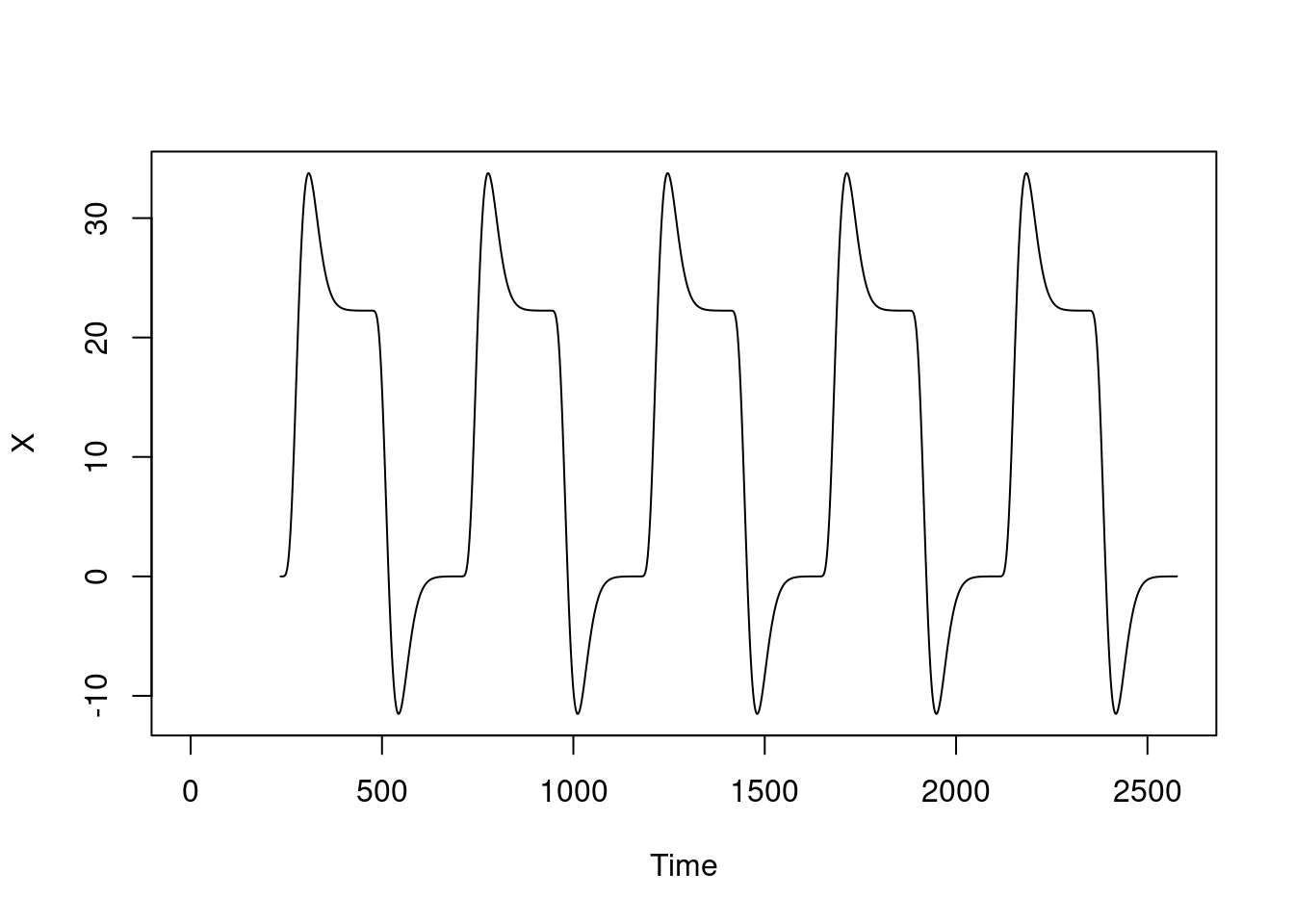 Para o comportamento da deoxy, espero o comportamento inverso (deoxy está negativamente correlacionada com a oxy).
Para o comportamento da deoxy, espero o comportamento inverso (deoxy está negativamente correlacionada com a oxy).
Estamos modelando de forma similar:
# Vetor que contem as estatisticas t dos betas
# do GLM para cada canal (nesse experimento são 20)
mapaToxy = array(0, 20)
mapaTdeoxy = array(0, 20)
for(canal in 1:20){
# ajuste do GLM para oxy
# o que antes eram todos os voxeis de fMRI, em NIRS são os 20 canais
# o modelo prevê a OXY de cada canal em funço da HRF convoluída da cond.
# dessa forma, ao ter cond =1 pra tarefa e 0 para repouso, o teste:
modelo = lm(OXY[1:2577, canal]~X)
mapaToxy[canal] = summary(modelo)$coef[2, 3] #estat. t
#ajuste do GLM para deoxy
modelo = lm(DEOXY[1:2577, canal]~X)
mapaTdeoxy[canal] = summary(modelo)$coef[2, 3] #estat. t
}O mapa é o resultado do teste estatístico em todos os canais.
modelo1 = lm(OXY[1:2577, 1]~X)
mapaToxy[canal] = summary(modelo1)$coef[2, 3] #estat. t
summary(modelo1) ##
## Call:
## lm(formula = OXY[1:2577, 1] ~ X)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.0008340 -0.0001565 -0.0000107 0.0001493 0.0009014
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -3.349e-05 6.535e-06 -5.124 3.24e-07 ***
## X 3.263e-06 3.686e-07 8.851 < 2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.0002464 on 2341 degrees of freedom
## (234 observations deleted due to missingness)
## Multiple R-squared: 0.03238, Adjusted R-squared: 0.03197
## F-statistic: 78.34 on 1 and 2341 DF, p-value: < 2.2e-16 #ajuste do GLM para deoxy
modelo2 = lm(DEOXY[1:2577, 1]~X)
mapaTdeoxy[canal] = summary(modelo2)$coef[2, 3]
summary(modelo2) #estat. t##
## Call:
## lm(formula = DEOXY[1:2577, 1] ~ X)
##
## Residuals:
## Min 1Q Median 3Q Max
## -5.997e-04 -1.231e-04 6.810e-06 1.172e-04 4.925e-04
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 6.990e-06 4.636e-06 1.508 0.1317
## X 4.553e-07 2.615e-07 1.741 0.0818 .
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.0001748 on 2341 degrees of freedom
## (234 observations deleted due to missingness)
## Multiple R-squared: 0.001293, Adjusted R-squared: 0.0008668
## F-statistic: 3.032 on 1 and 2341 DF, p-value: 0.08178Uma consideração do GLM é que os erros (resíduos) são independentes. Na prática, como a taxa de amostragem do NIRS é superior a do fMRI, o problema da autocorrelação dos resíduos é maior. Para mapas de ativação estatísticos, os procedimentos de pre-whitening, pre-colouring ou semi-paramétricos são necessários pois os valores de teste T do NIRS acabam com uma magnitude muito maior.
Coleta de fNIRS
Na aula o professor mostrou o equipamento NIRSport da empresa NIRx (Alemanha). Além da empresa NIRx, tem a Artinis e a Hitashi.
No software NIRx Star que se utiliza para fazer a calibração dos optodos e coleta do sinal.
Na calibração pode-se corrigir o ajuste da touca com:
- Verde: Sinal excelente
- Amarelo: Sinal bom
- Vermelho: Sinal ruim
- Branco: Canal morto
Ao iniciar a gravação, pode-se ver os sinais: vermelho a oxihemoblogina (HbO) e azul a desoxihemoglobina (Hb). Na onda, é possível ver o artefato cardíaco no sinal (mais forte na oxy pois vem das arteríolas). A deoxy se encontra nas vênulas, que é mais fraco. Uma vantagem em relação ao EEG é que como se trata de luz infravermelho e não pulsos elétricos, não há problemas de artefatos ocasionados por movimentos ou fala.
Pode-se utilizar para atividades de neurofeedback.
Na aula, foi mostrado com um voluntário um experimento de geração de números aleatórios, intercalando dois tipos de atividades:
- Contagem de 0-9
- Gerração de números aleatórios
Após a coleta, o professor mostrou como processa no NIRSLab.
- Carregar arquivo coletado
- Correção de triggers (marcação do experimento)
- Limpeza do sinal: Remoção de spikes e filtragem do sinal: escolha dos filtros. Na filtragem são filtrados comprimentos de onda e não a oxy e deoxy. A correção do movimento não é realizada no software
- Cômputo de estados hemodinâmicos: Estabelecer parâmetros do coeficiente de absorção de Beer-Lambert e cômputo de estados hemodinâmicos.
- Aplicação do GLM: Em SPM Level, pode-se escolher qual onda trabalhar. Em “Specific basis” escolhemos a HRF canônica, com a leitura dos triggers. O pre-colouring e pre-whitening também é possível adicionar nas configurações.
Nesse caso, o GLM para cada canal é:
e na visualização é possível ver cada
Para ver essas regiões são estatísticamente significantes, é necessário especificar o contraste (marcar com 1 e 0). É possível verificar por exemplo maior ativação no hemisfério oposto a mão que escreve.
No experimento sugere-se que as tarefas sejam alternadas por conta do artefato de inclinação. No desenho experimental, alterna-se as tarefas para garantir que a variação da hemodinâmica seja realmente da tarefa cognitiva.
Prova: Sobre NIRs.